Dans une cellule, il existe tout un cortège de protéines qui s'affaire autour de l'ADN pour pouvoir le lire, le protéger, le réparer… Parmi les différents acteurs impliqués dans ce travail, les nucléases, qui coupent l'ADN, sont indispensables notamment pour réparer des cassures. La nucléase humaine MRN, formée par l'association de deux protéines Mre11 et Rad50, est un acteur clé de la recombinaison homologue et de la méiose, deux processus qui utilisent des mécanismes de réparation des cassures.
Une nucléase verrouillée
MRN n'est pas spécifique aux mammifères. Elle est conservée, on la retrouve par exemple dans les bactéries (sous le doux patronyme de SbcC-SbcD). Chez les eucaryotes, la nucléase s'est équipée d'un verrou qui contrôle l'activation de l'enzyme : celle-ci n'est possible que durant certaines phases du cycle cellulaire (entrée en G2, lorsque l'ADN est dupliqué pour permettre le processus de recombinaison), lorsque le verrou est phosphorylé.
Ce verrou c'est CtIP, Sae2 chez la levure
Saccharomyces cerevisiae. C'est une protéine à la structure largement désordonnée, comme un long spaghetti. De tels objets moléculaires sont de véritables casse-têtes : leur structure tridimensionnelle est très difficile à déterminer (trop flexible, trop de configurations possibles, trop de phénomènes parasites comme leur oligomérisation durant les tentatives de cristallisation). En l'occurrence, alors que Sae2 a été découvert il y a près de 30 ans, personne n'a encore réussi à déterminer sa structure.
Un modèle structural renforcé par des expériences in vitro et in vivo
Dans une étude publiée dans
Molecular Cell, des équipes de l'I2BC, de l'institut Curie et de l'IRB (Bellinzona, Suisse) décryptent les mécanismes d'action de Sae2 sur MRN.
L’équipe
AMIG (Asssemblages moléculaires et Intégrité des Génomes) de l'I2BC a réussi à modéliser la structure du complexe MRN de levure grâce à l'algorithme AlphaFold2. De manière assez fascinante, le programme hésite lors de la modélisation entre deux états : inactif (auto-inhibé) et actif. Surtout, l'addition de Sae2 phosphorylée favorise la conformation active dans les prédictions d'AlphaFold2 : Sae2 déverrouillerait le complexe MRN par l'établissement d'un ensemble synergique d'interactions centrées sur la phosphorylation d'une sérine de Sae2.
Différents sets de mutations des partenaires protéiques ont alors été imaginés pour tester le modèle : certaines mutations devant déstabiliser le réseau d'interactions tel qu'il apparait dans les modèles prédits par AlphaFold2, d'autres combinant plusieurs mutations individuelles se compensant entre elles. Les équipes de l'Institut Curie et de l'IRB ont mené des expériences
in vitro (sur protéines recombinantes mutées et purifiées) et
in vivo (mesure du taux de recombinaison de cassures d'ADN, test de phénotypes méiotiques) qui valident le modèle structural obtenu avec AlphaFold2.
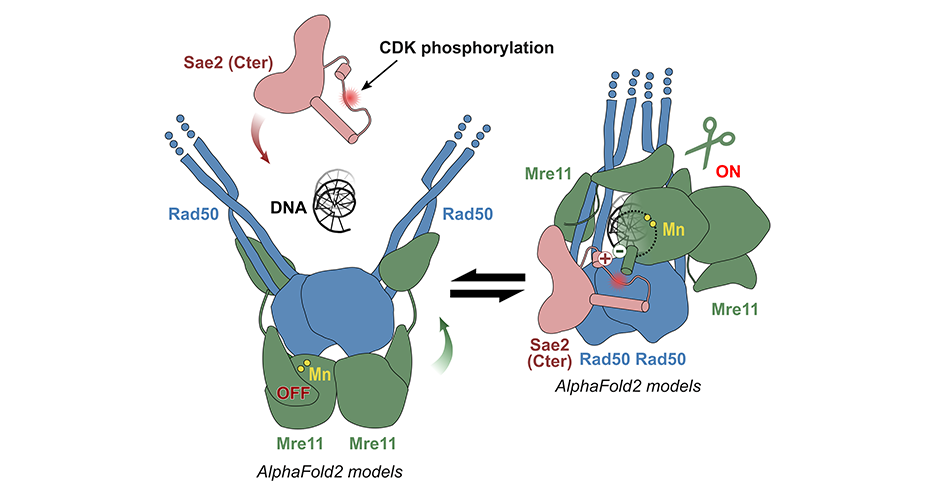
Modèles structuraux de la nucléase MRN (Mre11-Rad50) prédits par AlphaFold2 : à gauche, un modèle inactif (auto-inhibé) dans lequel Rad50 bloque le site actif de Mre11 ; à droite un modèle actif favorisé par la protéine Sae2 phosphorylée en S267. © R. Guerois / CEA
Les mécanismes décrits chez la levure sont-ils conservés chez l'Homme ?
Les séquences de Sae2 et CtIP ont fortement divergé l'une de l'autre au cours de l'évolution. Pour autant, dans une analyse évolutive, les chercheurs montrent que CtIP comme Sae2 est capable de maintenir la conformation active de Mre11-Rad50 mais avec un motif structural très différent.
Toutes ces données montrent comment une protéine en apparence désordonnée peut lever l'auto-inhibition d'une nucléase et ainsi contrôler l'aiguillage entre différentes voies de réparation, importantes chez l'Homme pour la progression dans le cycle cellulaire et la méiose.
Contact institut des sciences du vivant Frédéric-Joliot / I2BC :