L'épiderme héberge des cellules souches dotées de fonctions de renouvellement et de régénération bien documentées. Il constitue ainsi un modèle de choix pour décrypter les réseaux de molécules régulatrices qui contrôlent les caractéristiques et fonctionnalités des cellules souches des tissus humains.
Les recherches ont été longtemps focalisées sur la partie du génome codant le protéome, mais l'émergence du domaine des ARNs non codants augmente la complexité du vivier des molécules régulatrices potentiellement impliquées. Le développement d'approches bio-informatiques de bio-analyse prospective revêt une place centrale pour l'identification de pistes originales dans ce domaine.
C'est dans ce contexte que les chercheurs du laboratoire LGRK (iRCM) ont mis en œuvre dans une nouvelle étude publiée dans International Journal of Molecular Sciences un cadre méthodologique d'analyse de données biologique visant à apporter un nouvel éclairage sur deux jeux de données de séquençage ARN (RNA-seq) précédemment générés dans des études (1,2) pilotées par le même laboratoire. Ces études avaient mis en évidence les rôles de deux facteurs de transcription, KLF4 et MXD4/MAD4, dans le contrôle du caractère « cellule souche » ou « stemness » dans le lignage des kératinocytes épidermiques.
En utilisant des outils d'interférence par ARN pour moduler à la baisse l'expression des gènes codant pour KLF4 et MXD4/MAD4, les chercheurs ont observé un état de "stemness" accru des cellules, indiquant l'importance de ces gènes dans le maintien de l'état immature des cellules souches.
Le séquençage de l'ARN a permis de détecter des changements transcriptionnels significatifs associés à la modulation des gènes KLF4 et MXD4/MAD4, fournissant des éléments de compréhension détaillés sur les mécanismes moléculaires sous-jacents. L'approche informatique, en intégrant les données du génome codant (ARNm) et du génome non codant (ARN longs non codants), ainsi que des micro-ARN (miARN) validés, a mis en évidence des liens potentiels entre ces différents types de transcrits.
Les chercheurs ont construit grâce à une approche in silico des unités de régulation (régulons) constituées de transcrits codants et non codants, identifiant ainsi de nouveaux effecteurs potentiels de l'état "stemness" des cellules, ouvrant ainsi la voie à la sélection de candidats pro- « stemness » originaux. Ce vivier est ainsi disponible pour des approches expérimentales de validation fonctionnelle.
En intégrant des données fonctionnelles et des données obtenues par modélisation informatique, cette étude permet de dresser une représentation prospective de la complexité des transcrits régulant l'état de "stemness" des kératinocytes, et ouvre la voie à de nouvelles recherches et applications thérapeutiques.
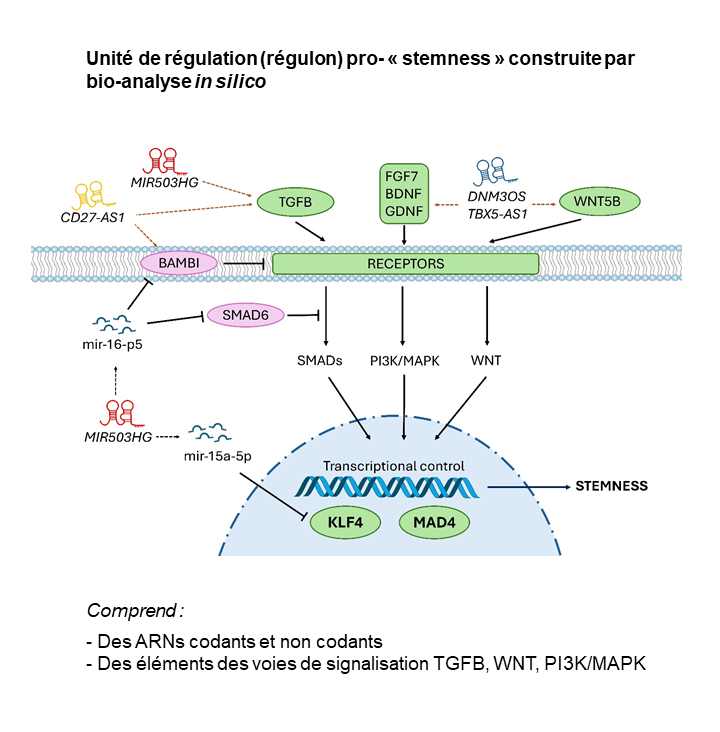
Unité de régulation pro-« stemness » construite par bio-analyse in silico
(crédit image N. Fortunel/LGRK)