Les protéines sont des molécules importantes du domaine vivant, car elles prennent en charge la plupart des fonctions biologiques. Leurs modifications enzymatiques, notamment l’addition ou la soustraction de divers groupements chimiques, permet de moduler leurs activités. C’est une des stratégies utilisées par les systèmes vivants pour s’adapter aux conditions qu’ils rencontrent. Afin de suivre ces changements notables, une équipe de l’Irig développe des méthodes protéomiques basées sur la spectrométrie de masse pour caractériser à large échelle les modifications des protéines.
Une des modifications fréquemment réalisées sur les protéines consiste à additionner ou à soustraire un groupement phosphate par des protéines
kinases et des phosphatases. Ces modifications, désignées
phosphorylation et déphosphorylation, permettent de réguler finement l’activité des protéines cibles. Il est donc important d’identifier quels sites des protéines sont modifiés et leurs niveaux de modification, en fonction des conditions physiopathologiques étudiées, afin de déterminer les processus moléculaires mis en jeu.
Ainsi, une équipe de l’Irig grâce à son expertise en biochimie, en sciences analytiques et en science des données, a développé une méthodologie permettant d’identifier et de quantifier simultanément des milliers de sites de phosphorylation de protéines dans des échantillons complexes. Par exemple, dans le cadre d’études sur la maladie de Rendu-Osler, cette méthode a permis récemment de découvrir de nouvelles voies de signalisation régulées par les protéines BMP9 et BMP10 dans les cellules endothéliales (FM Biosanté) [1]. Un autre exemple d’application de cette méthode, concerne la compréhension de mécanismes moléculaires utilisés par le virus de l’hépatite B pour pirater la machinerie cellulaire à son profit dans des hépatocytes primaires [2].
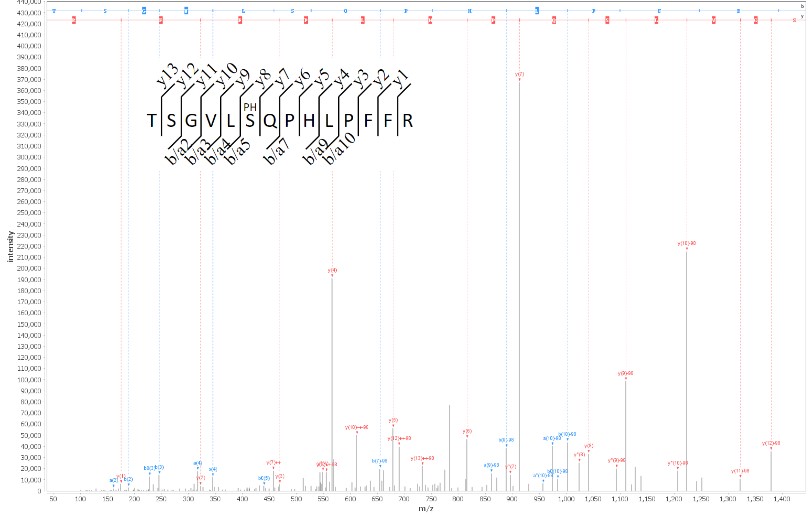
Figure : identification d’un site de phosphorylation par analyse protéomique basée sur la spectrométrie de masse. Après digestion des protéines de l’échantillon analysé, les peptides produits sont analysés par spectrométrie de masse et les spectres générés interprétés par des outils ad-hoc afin d’identifier leurs séquences en acides aminés et leurs modifications.
Les développements réalisés permettent aujourd’hui d’identifier et quantifier environ 10 000 sites de phosphorylation dans un protéome complexe en une seule analyse, permettant de mieux comprendre les mécanismes qui régulent les fonctionnements physiologiques et pathologiques des systèmes biologiques.

Photo : couplages nanoLC-MS (Credit CEA)
Une protéine
kinase est une enzyme catalysant le transfert d'un groupe phosphate d'une molécule donneuse à une protéine cible.
La
phosphorylation est l’addition d’un groupe phosphate à une protéine ou une petite molécule.
Financements
ANR (Agence Nationale de la Recherche) :
- ProFI (Proteomics French Infrastructure)
- GRAL
via Chemistry Biology Health Graduate School at University Grenoble Alpes
ANRS (Agence Nationale de Recherche sur le Sida et les hépatites virales) :