Dans certains mécanismes de réparation de l'ADN, recombinaison homologue et méiose, la protéine MRN joue un rôle clé. Cette nucléase s'est conservée au cours de l'évolution, présente chez tous les eucaryotes (organismes dont les cellules ont un noyau). Chez la levure et l'Homme, la MRN s'est équipée d'un verrou pour contrôler son activation : elle est active lorsque son verrou est phosphorylé. Or, cette phosphorylation, qui modifie les activités et interactions entre protéines, n'est possible que lors de la deuxième phase du cycle cellulaire, c'est-à-dire une fois que l'ADN est dupliqué pour permettre la recombinaison.
Pour décrypter ces mécanismes, les scientifiques s'intéressent à la levure Saccharomyces cerevisiae et au verrou Sae2 de sa nucléase MRN. Mais, la structure de cette protéine est tellement désordonnée qu'elle est un véritable casse-tête à résoudre. De fait, depuis sa découverte il y a trente ans, personne n'a pu déterminer la structure de Sae2. C'est ce que viennent de faire les chercheurs du CEA-Joliot, avec l'Institut Curie et l'Institut suisse de recherche en biomédecine.
Déverrouiller la nucléase en levant son auto-inhibition
Grâce à l'algorithme de Google Deepmind AlphaFold2, ils ont réussi à modéliser la structure de la nucléase de la levure. Si le programme hésite lors de la modélisation entre deux états, inactif (auto-inhibé) et actif, il montre surtout que l'addition de Sae2 phosphorylée permet de déverrouiller la MRN. Et cela, en créant un réseau d'interactions lors de la phosphorylation qui favorise la levée de son auto-inhibition.
Différents types de mutations des partenaires protéiques ont alors été imaginés pour tester le modèle. Certaines mutations devaient déstabiliser le réseau d'interactions tel qu'il apparait dans les modèles prédits par AlphaFold2. D'autres devaient combiner plusieurs mutations individuelles se compensant entre elles. Le défi est réussi car les expériences menées in vitro (sur des protéines recombinantes mutées et purifiées) ainsi qu'in vivo (par exemple, mesure du taux de recombinaison de cassures d'ADN) valident le modèle structural obtenu avec AlphaFold2.
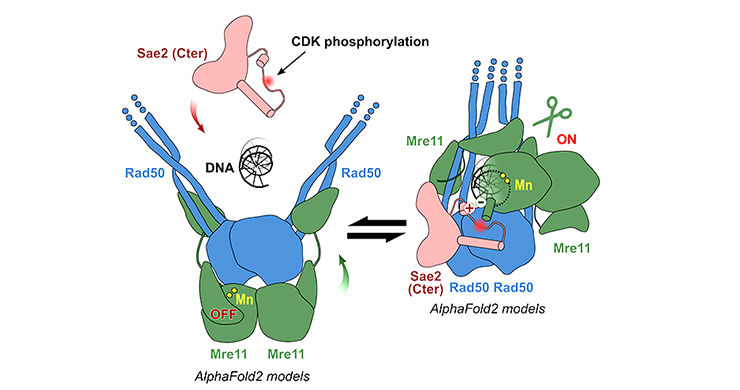
Modèles structuraux de la nucléase MRN (Mre11-Rad50) prédits par AlphaFold2 : à gauche, un modèle inactif (auto-inhibé) dans lequel Rad50 bloque le site actif de Mre11 ; à droite un modèle actif favorisé par la protéine Sae2 phosphorylée en S267. © R. Guerois / CEA
Un mécanisme conservé différemment chez l'homme
Si les chercheurs savent que les séquences de Sae2 de la levure et de CtIP, son homologue chez l'Homme, ont fortement divergé l'une de l'autre au cours de l'évolution, ils montrent toutefois que les deux sont capables de maintenir la conformation active de la MRN, mais avec un motif structural très différent.
« Toutes ces données montrent comment une protéine en apparence désordonnée peut lever l'auto-inhibition d'une nucléase et ainsi contrôler l'aiguillage entre différentes voies de réparation, importantes chez l'Homme pour la progression dans le cycle cellulaire et la méiose », conclut Raphaël Guérois du CEA-Joliot.